Un equipo de investigación ha hecho avances significativos en la generación de enzimas con el uso de inteligencia artificial generativa, mediante modelos de lenguaje de proteínas (pLMs) que amplían la diversidad natural de estas biomoléculas de alto valor. Esta estrategia no solo diversifica las enzimas disponibles, sino que también busca mejorar su estabilidad, especificidad y efectividad en células humanas, un aspecto esencial en la biotecnología y la medicina.
La empresa Metagenomi está a la vanguardia en el desarrollo de terapias curativas utilizando enzimas de edición genética CRISPR. Su enfoque implica la utilización de una base de datos extensa de enzimas naturales (MGXdb) para identificar candidatos prometedores y entrenar modelos de lenguaje de proteínas. Estos modelos permiten generar variantes adicionales de ciertas clases de enzimas, que luego son filtradas utilizando flujos de trabajo de modelos múltiples para predecir características clave y facilitar campañas de ingeniería de proteínas.
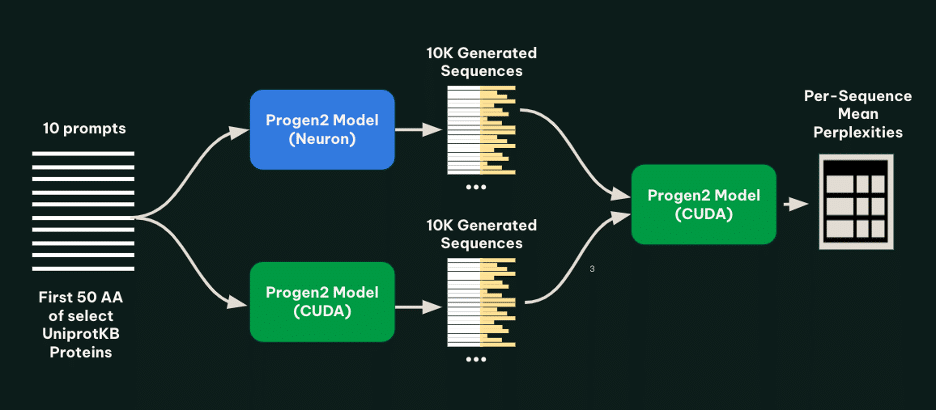
En un reciente artículo, el equipo de Metagenomi compartió métodos para reducir los costos asociados a la generación de proteínas mediante el uso del modelo Progen2 en instancias AWS Inferentia, logrando una disminución de hasta el 56% en los costos de generación de variantes en comparación con otras tecnologías basadas en GPU. Esta implementación, llevada a cabo en colaboración con Tennex y el equipo de AWS Neuron, permite una generación de enzimas a gran escala, algo que es crucial para la diversificación dentro de las clases de enzimas de alto valor.
El uso de instancias EC2 Inf2 facilitó la escala masiva de inferencias a través de AWS Batch, un servicio que permite la ejecución eficiente de cientos de miles de trabajos computacionales. Esta infraestructura no solo optimiza el tiempo de generación, sino que también asegura que se minimice el gasto, una consideración vital en la investigación y desarrollo de biotecnología.
Además, el equipo llevó a cabo pruebas que comparaban diferentes métodos de generación, demostrando que las modificaciones implementadas no comprometían la precisión de los modelos. Las pruebas involucraron la generación de miles de secuencias de proteínas, estableciendo que las instancias Spot de EC2 Inf2 eran significativamente más económicas.
Con la meta de generar millones de proteínas, Metagenomi ha estado utilizando su acceso a una base de datos amplia de metagenómica y ha adaptado su modelo para maximizar la eficiencia y la eficacia en la generación de enzimas. Este proyecto no solo es un avance en el diseño de proteínas, sino que también representa un hito en la reducción de costos en biotecnología, lo que podría abrir nuevas avenidas para la generación de proteínas de alto valor que podrían transformar diversos sectores, desde la farmacéutica hasta la agricultura.
vía: AWS machine learning blog